AI ska kartlägga proteiner och öka förståelsen för hur celler fungerar
Artificiell intelligens (AI) och djupinlärning blir allt viktigare för att kartlägga proteinerna i våra celler och hur de interagerar med varandra. I ett nytt projekt ska forskarna utveckla verktyg för att öka förståelsen hur celler fungerar.

Inom livsvetenskaperna (life sciences) har de senaste decennierna skett en stor omvandling. Från att ha varit ”datafattigt” finns nu där stor tillgång på forskningsdata. De senaste åren har det även skett en revolution inom utvecklingen av metoder för djupinlärning som gör det möjligt att använda stora datamängder.
Genom att kombinera de senaste framstegen inom dessa två områden skapas möjligheter att revolutionera medicinska behandlingar. Redan nu finns exempel på sådana framsteg, främst inom forskningen om att förutsäga proteinstrukturer. Djupinlärningsverktyget AlphaFold från DeepMind är det kanske tydligaste exemplet. Sommaren 2021 publicerades två artiklar om AlphaFold och DeepMind släppte koden fritt till alla forskare. Detta satte igång en process där hundratals forskare nu har använt koden på olika sätt och resulterat i många vetenskapliga publiceringar.
30 miljoner till forskning om celler
En forskargrupp under ledning av Arne Elofsson, professor i bioinformatik vid Stockholms universitet och verksam vid Institutionen för biokemi och biofysik och SciLifeLab, tilldelas nu ett forskningsanslag på 30 miljoner av Knut och Alice Wallenbergs Stiftelse för forskning om cellens molekylära komponenter. I gruppen ingår även forskare vid Karolinska Institutet och KTH. Forskarna ska använda djupinlärningsmetoder för att identifiera alla proteinformer som finns i en cell, samt deras interaktioner med varandra. De 20 000 gener som finns hos oss människor ger upphov till många fler proteinformer genom olika slags modifieringar. Exakt hur många proteinformer som finns och vilka som är viktiga är till stora delar fortfarande okänt.
– Inom projektet hoppas vi kunna kartlägga de flesta protein-interaktioner som finns i en mänsklig cell. För att uppnå detta kommer vi att behöva utveckla nya metoder baserade på artificiell intelligens och utnyttja ny information från storskaliga experiment. Vi hoppas att projektet kommer att leda till utveckling av nya verktyg som kan användas av alla, samt till en ökad förståelse för hur en cell fungerar, säger Arne Elofsson.
Än mer exakta modeller av proteinkomplex
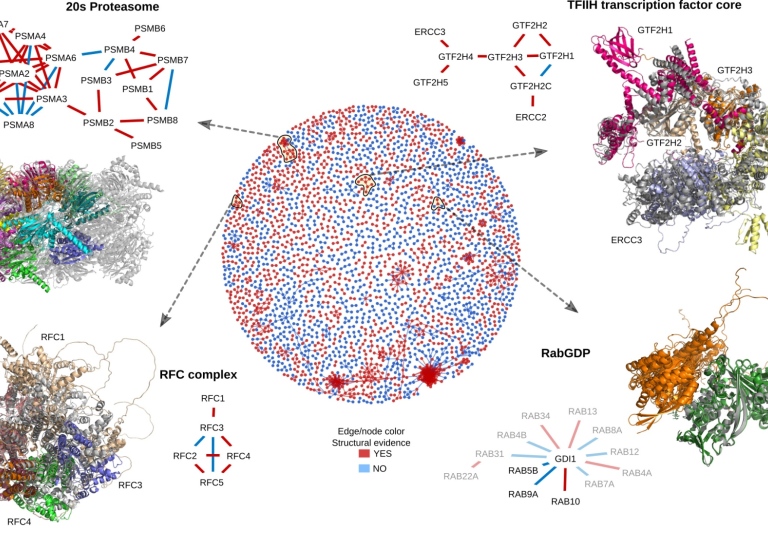
Han och hans kolleger har redan använt sig av AlphaFold och utvecklat nya metoder för att studera interaktioner mellan proteiner och förbättrat metoderna för att förutsäga strukturen av stora proteinkomplex. De proteinformer de hoppas identifiera ska nu användas för att bygga än mer exakta modeller av proteinkomplex. För att testa förutsägningar och skapa mer experimentell data kommer de även att använda sig av masspektrometri. Det är en teknik som snabbt kan ge information om hur proteiner veckar sig och interagerar.
För att kunna modellera alla proteiner och dess interaktioner i en mänsklig cell behövs dock ännu bättre djupinlärningsmetoder, mer experimentell data och tillgång till mycket datakapacitet. Här kommer forskarna att utnyttja den snabba utvecklingen som skett av öppen mjukvara. Forskargruppen har redan utvecklat egen öppen mjukvara och kommer att fortsätta att göra det.
Läs även artikeln Fyra stora Wallenberganslag till Stockholms universitet
Senast uppdaterad: 18 oktober 2022
Sidansvarig: Kommunikationsavdelningen

